Dr. Dr. Daniel Hübschmann möchte Krebstherapien präziser machen. Dafür sucht der Bioinformatiker und Mediziner am Deutschen Konsortium für Translationale Krebsforschung (DKTK) in genomischen Daten nach Informationen darüber, wo genau die Schwachstelle eines Tumors liegt.
Krebserkrankungen werden bisher nach dem Gewebe klassifiziert, in dem sie vorkommen. Mediziner wie Laien sprechen daher von Brustkrebs, Lungenkrebs oder Darmkrebs. Dabei kann Krebs ganz unterschiedliche molekulare Ursachen haben. Für eine effektive Behandlung muss man daher zunächst herausfinden, welche Gene mutiert und dafür verantwortlich sind, dass Zellen entarten und sich unkontrolliert teilen.
Dafür sequenziert man das Erbgut der Tumorzellen. Mit dem sogenannten Next Generation Sequencing ist das zunehmend schneller und kostengünstiger möglich. Doch die eigentliche Arbeit beginnt erst danach: In dem schier undurchdringlichen Datendschungel liegen wahre Schätze versteckt – Hinweise darauf, wo die Krebszellen angreifbar sind und mit welchen Medikamenten man ihnen beikommen könnte. Nur muss man diese Informationen erst einmal finden.
An dieser Aufgabe arbeitet Daniel Hübschmann, Physiker, Mathematiker und Arzt mit Doktortiteln in Medizin und Biowissenschaften, inzwischen hauptberuflich als Bioinformatiker. Er leitet zwei Arbeitsgruppen: „Mustererkennung und digitale Medizin“, angesiedelt am Deutschen Krebsforschungszentrum (DKFZ) und am Heidelberger Institut für Stammzelltechnologie und Experimentelle Medizin (HI-STEM gGmbH), sowie „Computergestützte Onkologie“ – am DKFZ, dem Kernzentrum des Deutschen Konsortiums für Translationale Krebsforschung (DKTK) und dem Nationalen Centrum für Tumorerkrankungen Heidelberg (NCT Heidelberg) beheimatet.
Nach seinem Studium war Hübschmann zunächst drei Jahre lang als Arzt auf der Kinderkrebs-Station des Universitätsklinikums Heidelberg tätig. „Das Leid, das man dort in einzelnen Fällen erlebt, ist wirklich riesig“, sagt Hübschmann. So wuchs seine Motivation, mithilfe von Forschung die Therapien gegen Krebs zu verbessern. Mit viel Rechenpower sucht Hübschmann heute im Rahmen des im DKTK Joint-Funding-Förderprogramms finanzierten Projekts „MARRIAGE“ nach Mustern im Erbgut von Krebszellen, die darauf hindeuten, dass ein wichtiges zelleigenes Reparatursystem gestört ist. An MARRIAGE sind auch Wissenschaftlerinnen und Wissenschaftler von den DKTK-Partnerstandorten Frankfurt/Mainz und Dresden beteiligt. Sie alle wollen besser erkennen, ob die betroffenen Krebszellen besonders gut von einer bestimmten Medikamentenkombination vernichtet werden können. Dann könnte man Patientinnen und Patienten eine auf das Genom ihres Tumors maßgeschneiderte Therapie anbieten.
Denn wenn Zellen sich teilen, müssen sie zunächst ihre DNA verdoppeln. Dabei passieren ihnen ständig Fehler: Mal wird aus Versehen ein falscher DNA-Baustein eingefügt oder es fehlt einer, mal bricht ein DNA-Strang. Dank ausgeklügelter Reparaturmechanismen können diese Defekte in den meisten Fällen wieder behoben werden. „Ein besonders effizienter Prozess ist die sogenannte ‚Homologe Rekombination‘“, sagt Daniel Hübschmann. Sie funktioniert, weil alle Chromosomen im Zellkern doppelt vorliegen und somit jedes Chromosom einen Zwilling hat: Die Zelle nutzt als Vorlage für die Reparatur des defekten Chromosoms die korrekte Information des unbeschädigten, homologen Chromosoms.
Angriffstor für spezielle Therapie
Auch Krebszellen, die sich besonders rasant vermehren, sind auf solche Reparatursysteme angewiesen. Wenn bei ihnen die Homologe Rekombination (HR) nicht mehr funktioniert, öffnet sich dadurch ein Angriffstor für eine spezielle Kombinationstherapie: Die Patientinnen und Patienten erhalten zunächst ein Chemotherapeutikum, das zahlreiche Schäden im Erbgut der Krebszellen verursacht. Zusätzlich nehmen sie Medikamente namens PARP-Inhibitoren, die auch einen zweiten Reparaturmechanismus behindern. Die Krebszellen können dann die Schäden in ihrem Erbgut nicht mehr ausbessern und sterben schließlich ab. Doch bei welchen Patientinnen und Patienten die HR tatsächlich defekt ist, ist bisher schwer zu identifizieren.
„Noch sind wir für manche Sachen blind“, sagt Hübschmann. Denn bisher sind nicht einmal alle Gene bekannt, die an der Homologen Rekombination beteiligt sind. Zudem führt die Architektur der Chromosomen dazu, dass die Gen-Sequenzierung gerade in den kritischen Bereichen, wo bereits bekannte, wichtige Gene für die HR liegen, keine validen Ergebnisse liefert. Als letztes Problem kommt hinzu, dass Gene nicht nur durch Mutationen ihre Funktion verlieren, sondern auch über sogenannte epigenetische Veränderungen abgeschaltet werden können. Bekanntestes Beispiel dafür sind Methylgruppen, die an die DNA angehängt werden können und somit Gene deaktivieren. Solche epigenetischen Veränderungen lassen sich jedoch nicht allein mithilfe einer DNA-Sequenzierung aufspüren, sondern erfordern zusätzliche umfangreiche Analysen. „Es gibt also ganz viele Fälle, wo die HR in den Krebszellen defekt ist, uns das aber nicht auffällt“, sagt Hübschmann.
HOMOLOGE
REKOMBINATION
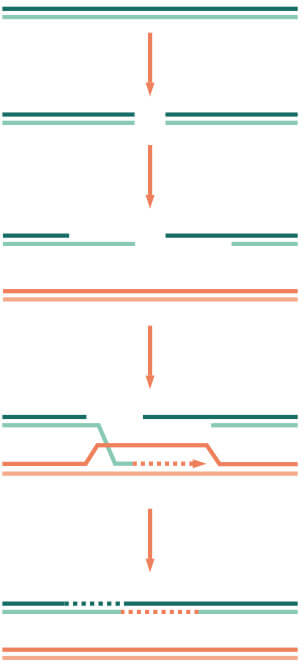
Mithilfe der homologen Rekombination (HR) können DNA-Doppelstrangbrüche repariert werden: Nachdem an der Bruchstelle Überhänge produziert wurden, dient ein homologer (gleichartiger) DNA-Abschnitt als Vorlage, um die Sequenz identisch wiederherzustellen.
WENN ZELLEN SICH TEILEN, MÜSSEN SIE ZUNÄCHST IHRE DNA VERDOPPELN. DABEI PASSIEREN IHNEN STÄNDIG FEHLER.
Hübschmanns Ziel ist es, die Homologe Rekombination besser zu verstehen und anhand der genomischen Daten schneller und zielgerichteter herauszufinden, bei welchen Krebszellen dieser Vorgang bereits nicht mehr funktioniert. Gemeinsam mit Kollegen hat er deshalb den sogenannten TOP-ARTS-core entwickelt – einen komplexen Biomarker, der anhand einer Analyse des gesamten Genoms abschätzt, ob das HR-System defekt ist. Patientinnen und Patienten, bei denen das zutrifft, können in eine klinische Studie eingeschlossen werden, ein zusammen mit mehreren Kollegen am NCT Heidelberg entwickeltes sogenanntes Investigator Initiated Trial. Sie werden in eine von zwei Gruppen eingeteilt: Eine Gruppe erhält die oben beschriebene Kombitherapie. Die andere wird gemäß der derzeitigen Standardtherapie behandelt. 102 Patienten soll die Kohorte schlussendlich umfassen, etwa 60 Prozent sind bereits rekrutiert. Da das HR-System in allen Krebsarten defekt sein kann, ist die Studie auch nicht auf einzelne Tumorarten begrenzt.
Das MARRIAGE-Projekt bildet die umfassende Begleitforschung zu dieser TOP-ART-Studie. Das Team am DKTK-Partnerstandort Dresden arbeitet daran, einzelne Tumorzellen der Patientinnen und Patienten zu kultivieren. Wenn das gelänge, wäre es ein mächtiges Instrument für die Medikamententestung. Am DKTK-Partnerstandort Frankfurt/Mainz spezialisiert man sich auf die Erkennung spezieller Resistenzmechanismen. Und Hübschmanns Team in Heidelberg arbeitet derweil am TOP-ART 2.0, einem noch spezifischeren und sensitiveren Biomarker. All diese Ansätze sollen die Therapie von bösartigen Tumoren individueller, zielgerichteter und effizienter gestalten – damit Patientinnen und Patienten noch besser behandelt werden können und größere Chancen auf Heilung bekommen.
NOCH SIND WIR FÜR MANCHE SACHEN BLIND.

